生命科学与技术学院薛剑煌团队建立5gmC精确测序方法并揭示其在莱茵衣藻中的分布与功能,成果发表于《核酸研究》
来源:生命科学与技术学院
时间:2025-09-28 浏览:
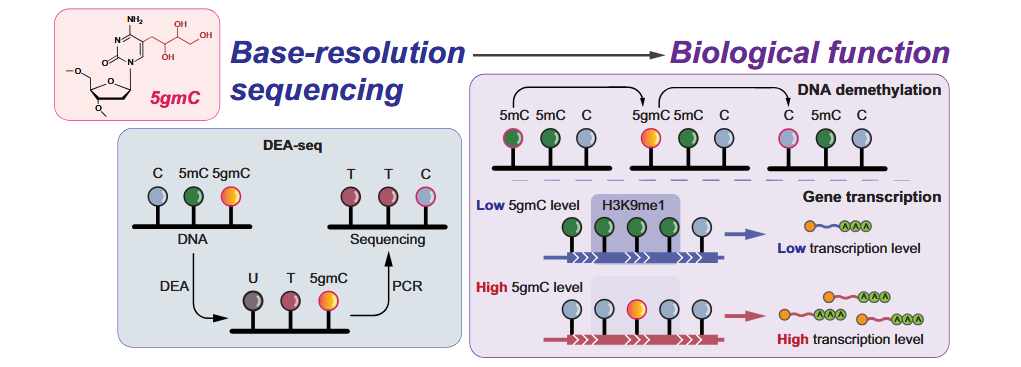
5-甘油基-甲基胞嘧啶(5gmC)是在模式生物莱茵衣藻(Chlamydomonas reinhardtii)中发现的一种新型DNA碱基修饰。与哺乳动物中TET家族蛋白将5-甲基胞嘧啶(5mC)逐步氧化为5-羟甲基胞嘧啶(5hmC)、5-醛基胞嘧啶(5fC)及5-羧基胞嘧啶(5caC)不同,衣藻中的TET同源蛋白CMD1以维生素C为辅因子,直接在5mC的甲基碳上引入甘油基,形成独特的5-gmC修饰。5gmC不仅可以参与DNA去甲基化过程,还可能作为一种新型表观遗传标记发挥作用,但其在基因组中的分布与功能还缺乏相应的研究。
9月25日,同济大学生命科学与技术学院薛剑煌课题组在《核酸研究》(Nucleic Acids Research)杂志上发表题为“Base-resolution profiling of 5-glyceryl-methylcytosine in Chlamydomonas reinhardtii via deaminase-assisted sequencing”的文章,首次在单碱基分辨率下揭示了莱茵衣藻中5gmC的全基因组分布特征。该研究建立了一种高效、低损伤的5gmC检测方法“DEA-seq”,发现5gmC可以作为一种独立的表观遗传标记,直接参与基因转录调控过程。

研究团队利用商品化DNA脱氨酶混合物(DEA)将DNA上的C和5mC脱氨为U或T,而5gmC因带有较大的甘油基可以抵抗DEA的脱氨作用,从而被保留为C。利用这个原理所建立的“DEA-seq”技术仅需一步反应就可以实现对5gmC的精确检测,不仅操作简便,且反应条件温和,因此所需DNA量少,适用于微量样本甚至单细胞测序。该方法也体现了使用单一脱氨酶区分不同DNA修饰的优越性与可靠性,但也强调了发现不同特性的脱氨酶与相应应用场景的重要性。
利用DEA-seq对两种常用莱茵衣藻藻株(CC125和CC5325)进行全基因组分析,研究团队发现5mC主要富集在CpG位点和启动子区域,与基因沉默相关;5gmC则偏好位于CHH位点(H为A、C或T),尤其富集于基因内含子区域,与基因活跃转录呈正相关。更值得注意的是,超过一半的5gmC位点与5mC位点互不重叠,而在其余重叠位点中5gmC与5mC水平呈负相关,提示5gmC可能作为5mC去甲基化的中间产物,同时也可能作为稳定的表观标记独立调控基因表达。功能分析显示,含有5gmC的基因多参与代谢和生物合成通路,尤其在光合作用相关基因中富集。研究还发现,5mC与抑制性组蛋白标记H3K9me1协同作用,共同抑制基因表达;而5gmC则很少与这些组蛋白修饰共定位,进一步支持其不同于5mC的转录调控功能。该研究为理解绿藻及其他生物中DNA修饰的多样性提供了重要资源。DEA-seq技术也可以被用于在其他物种中对胞嘧啶的复杂修饰进行检测。
同济大学生命科学与技术学院博士后王繁晨、博士研究生何宝丹为论文共同第一作者,生命科学与技术学院薛剑煌教授为论文通讯作者。研究工作还得到了中国科学院分子细胞科学卓越创新中心徐国良院士以及中国科学院水生生物研究所黄开耀研究员及研究团队的大力支持。该研究获得国家重点研发计划、国家自然科学基金、上海市自然科学基金和同济大学与附属同济医院启动经费等项目的资助。
论文链接:https://academic.oup.com/nar/article/53/18/gkaf955/8262767