生命科学与技术学院张勇课题组构建染色质调控基础模型ChromBERT,成果发表于《细胞基因组学》
来源:生命科学与技术学院
时间:2026-01-29 浏览:
转录调控因子、顺式调控元件与靶基因之间的相互作用共同构成转录调控网络,并在很大程度上决定细胞类型特异的基因表达程序。理解转录调控因子如何在特定基因组区域协同作用,以及这种调控结构如何随细胞类型、发育阶段或外界刺激而变化,是解析基因表达调控机制的关键问题。ENCODE等计划已积累了大量转录调控相关组学数据,但迄今为止,在“转录调控因子×细胞类型”这一组合空间上的数据覆盖度仍然极低,导致多数细胞语境下的转录调控规律缺乏直接数据证据。近年来,预训练基础模型在基因组学任务中显示出良好的表征学习能力:通过在大规模数据上进行预训练学习通用特征,再通过微调适配特定任务,有望在一定程度上缓解数据稀缺与跨细胞状态泛化的挑战。
1月26日,生命科学与技术学院张勇教授课题组在《细胞基因组学》(Cell Genomics)在线发表了题为“ChromBERT:A foundation model for learning interpretable representations for context-specific transcriptional regulatory networks”的文章,构建并发布了首个解码和可解释性地表征染色质上转录调控网络的基础模型ChromBERT。ChromBERT通过在大量人类ChIP-seq数据上进行预训练,学习到了全基因组位点特异性的调控因子互作语法,从而突破了依赖特定细胞类型数据的局限。通过轻量级微调,该模型能将这种通用的调控知识有效迁移至其它细胞语境。研究人员可利用ChromBERT模型高效地表征特定生物学场景下的转录调控网络,同时获得可解释的关键调控因子的线索。

现有的基因组基础模型大多聚焦于DNA线性序列,试图从碱基排列中学习通用规律。与这些模型不同,ChromBERT将转录调控因子在全基因组上的ChIP-seq定位信号作为模型输入,并将其视为一种可学习的协同调控模式。在该框架下,每一个基因组区域被表征为由多种调控因子共同定位所定义的组合状态;ChromBERT的核心任务是学习这些因子在不同基因组区域的共定位规律与协同作用结构。为此,团队构建了训练语料库:覆盖了人类991个转录调控因子、76种组蛋白修饰及染色质开放性数据,共计6391个高质量数据。这种跨细胞类型的数据池化策略在一定程度上提高了输入多样性,使模型能够学习更广泛的协同定位模式。ChromBERT采用掩码学习策略进行预训练:将全基因组划分为超过200万个1kb区域,并在训练中随机遮蔽部分因子的结合状态,促使模型根据其余因子的上下文组合模式推断被遮蔽信息。ChromBERT将每个因子在对应基因组区域上的协同作用语法压缩为高维特征嵌入,从而可以方便地对调控特征进行多角度分析,例如区分不同基因组区域的调控差异、比较同一因子在不同区域的上下文差异,以及刻画同一区域多因子协同作用等。
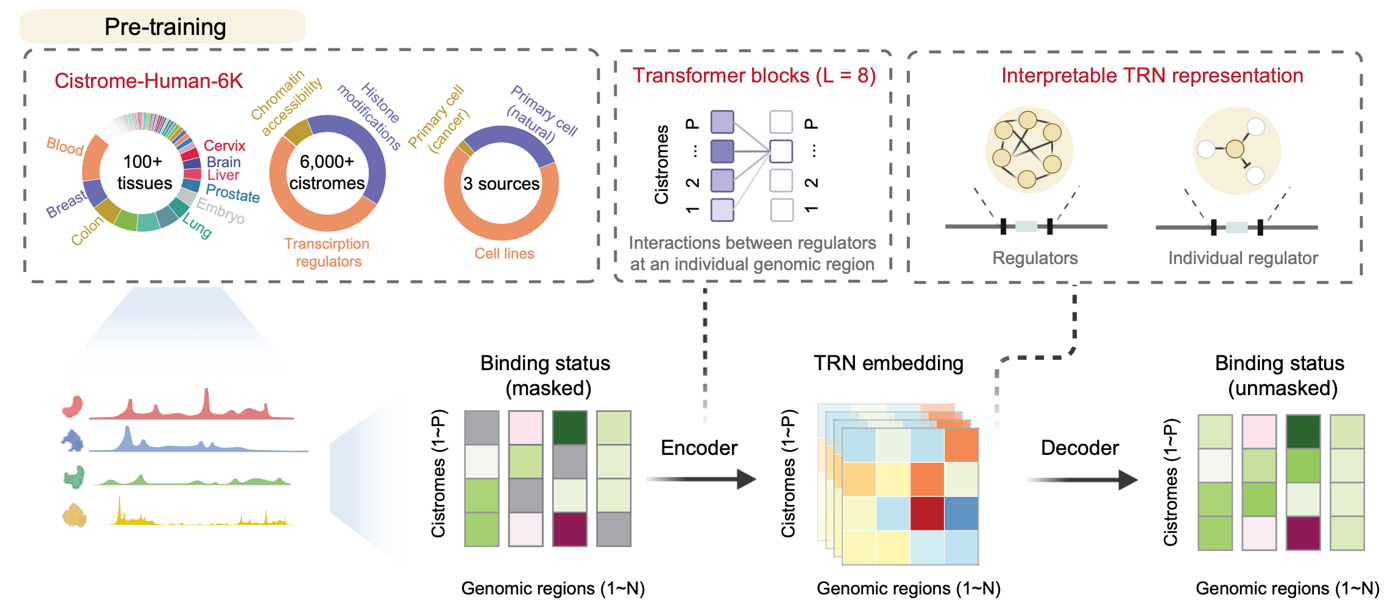
ChromBERT预训练
在预训练的基础上,通过微调ChromBERT模型可将通用的调控知识迁移到不同类型的生物学任务中:采用提示增强微调策略,模型能够对缺失的转录调控因子结合图谱进行高质量预测,并在跨细胞类型乃至单细胞等语境中保持良好的预测性能,表明其具有较好的鲁棒性与泛化能力;针对特定细胞类型或细胞状态转变过程进行任务特异性微调时,模型可结合细胞的转录组或染色质开放性数据对转录调控网络表征进行适配,从而有效推断特定细胞情境下关键调控因子的潜在功能。
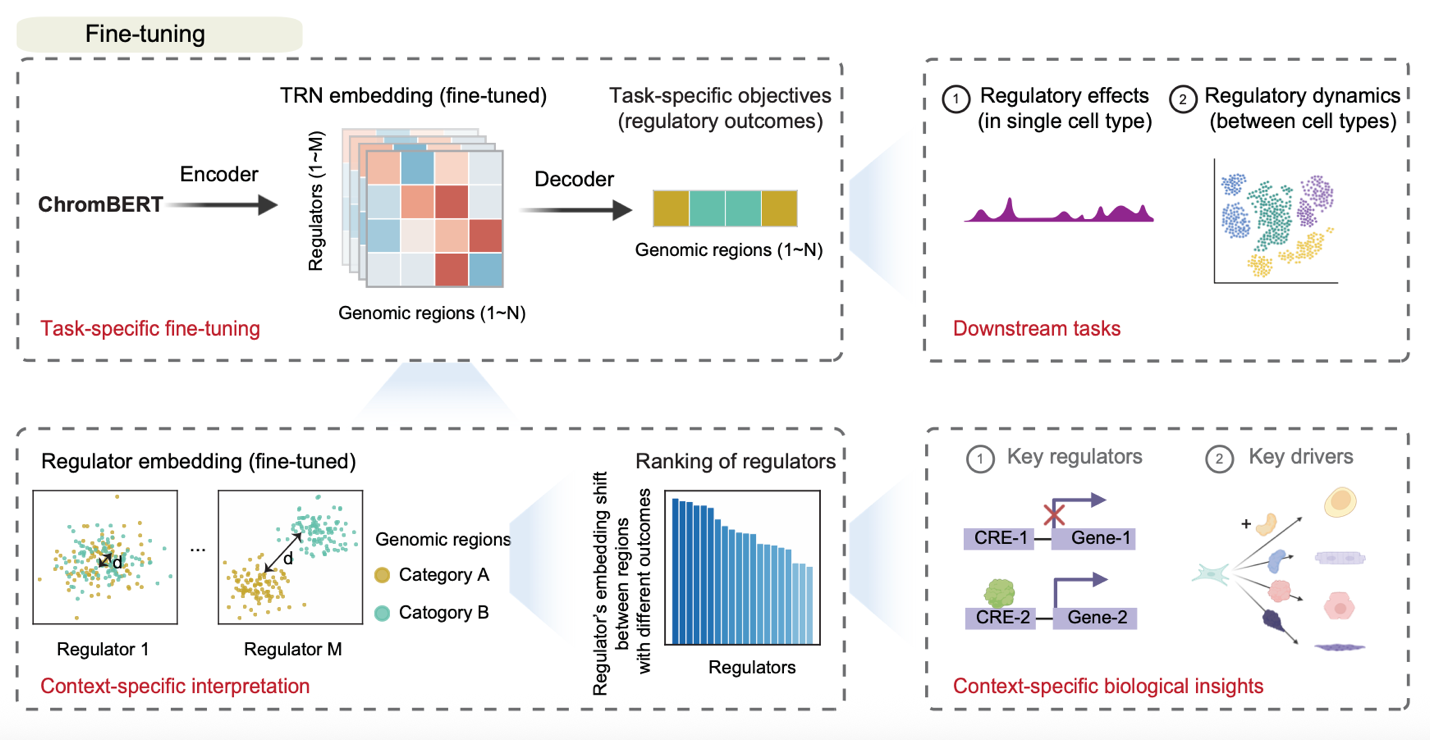
ChromBERT在特定细胞类型或细胞状态转变过程中的微调
同济大学生命科学与技术学院副教授余招伟、博士生杨东旭、陈倩倩和张雨轩为论文共同第一作者。同济大学生命科学与技术学院张勇教授为论文通讯作者。该研究工作得到了国家自然科学基金、上海市科委关键技术研发计划、科技部国家重点研发计划等项目的支持。
论文链接:https://www.cell.com/cell-genomics/fulltext/S2666-979X(25)00386-6