生命科学与技术学院史偈君团队开发三代测序检测RNA修饰的算法基准平台,成果发表于《自然·方法》
来源:生命科学与技术学院
时间:2025-12-12 浏览:
RNA修饰是“表观转录组学”的核心研究对象,广泛参与RNA稳定性、剪接和翻译等生物学过程,精准调控RNA命运。牛津纳米孔技术(ONT)推出了直接RNA测序方案(DRS),无需扩增直接对RNA分子进行测序,通过捕捉其穿过纳米孔时产生的电信号差异,并借助计算建模区分不同修饰类型,从而实现在单次实验中同时检测多种RNA修饰。随着该技术的应用,大量用于解析DRS数据中修饰信号的算法不断涌现。然而,这些算法的性能究竟如何?能否准确区分不同的修饰类型?面对DRS技术的持续迭代,计算工具应如何适应?这些问题始终悬而未决。
12月10日,同济大学生命科学与技术学院史偈君教授团队在国际知名期刊《自然·方法》(Nature Methods)上发表了题为“Systematic Evaluation of Computational Tools for Multitype RNA Modification Detection Using Nanopore Direct RNA Sequencing”的研究论文。该研究构建了高质量、单碱基分辨率的基准数据集作为“金标准”,并从准确性、生物学合理性、跨样本泛化能力及计算效率四个维度出发,设置了十余项评估指标,对86种基于DRS技术的RNA修饰检测算法进行了迄今为止最系统全面的“大比武”。研究涵盖m6A,假尿嘧啶(Ψ),m5C,A-to-I编辑,m1A和m7G这六种重要RNA修饰,不仅揭示了现有工具的性能差异与局限,还通过大规模重训练,展示了模型的迁移能力。
该研究的主要发现包括模型重训练策略显著提升检测性能:仅使用体外转录(IVT)RNA训练的工具在应用于真实生物样本时表现不佳。而将两者结合进行重训练,能极大提升工具的预测准确性和跨数据集泛化能力,尤其对Ψ、m5C和A-to-I等非m6A修饰效果明显。m6A检测工具整体表现优异,非m6A工具仍面临挑战:Dorado和SingleMod模型在m6A的定性和定量分析中均表现突出。然而大多数非m6A修饰检测工具在定量准确性和跨样本泛化方面仍明显不足。生物学合理性是重要试金石:理想的检测工具不仅需具备高准确性,其预测结果也应符合已知的生物学规律。研究发现,部分工具所预测的修饰位点分布与真实分布存在偏差。此外,m6Anet模型凭借其多示例学习(MIL)模块的合理设计,在区分野生型与酶敲除样本方面表现优异,凸显了算法设计对提升生物学解释力的重要性。工具难以区分相同碱基上的不同修饰:研究首次系统揭示,当前工具在单碱基分辨率下仍难以可靠区分发生在同一碱基上的不同修饰(如同样位于腺苷的m6A,m1A和A-to-I编辑),易导致“模糊预测”,是未来算法优化的重要方向。重训练模型可适配DRS技术迭代:随着DRS技术从RNA002升级至RNA004,测序通量得到提升,但电信号特征的变化使旧版算法无法直接迁移。研究提出的重训练模型可有效适配RNA004数据,缓解了新版本数据分析工具短缺的现状。
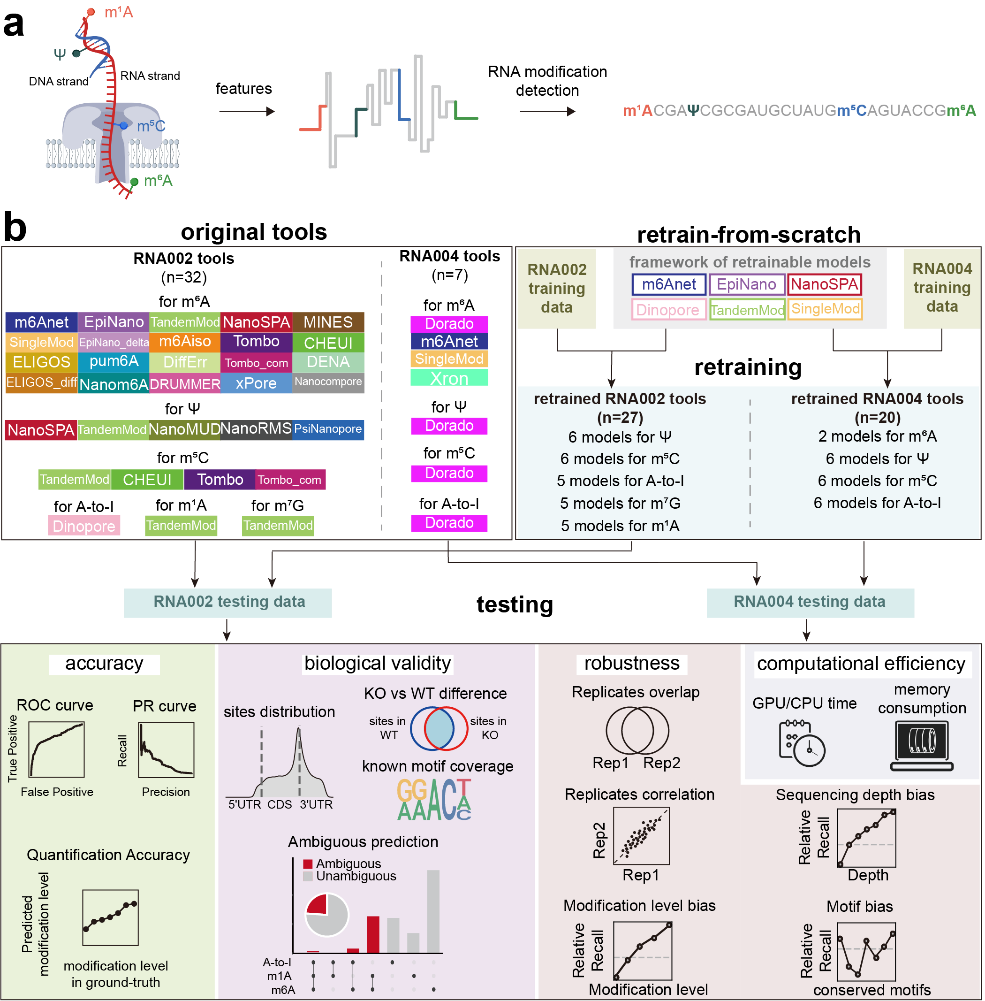
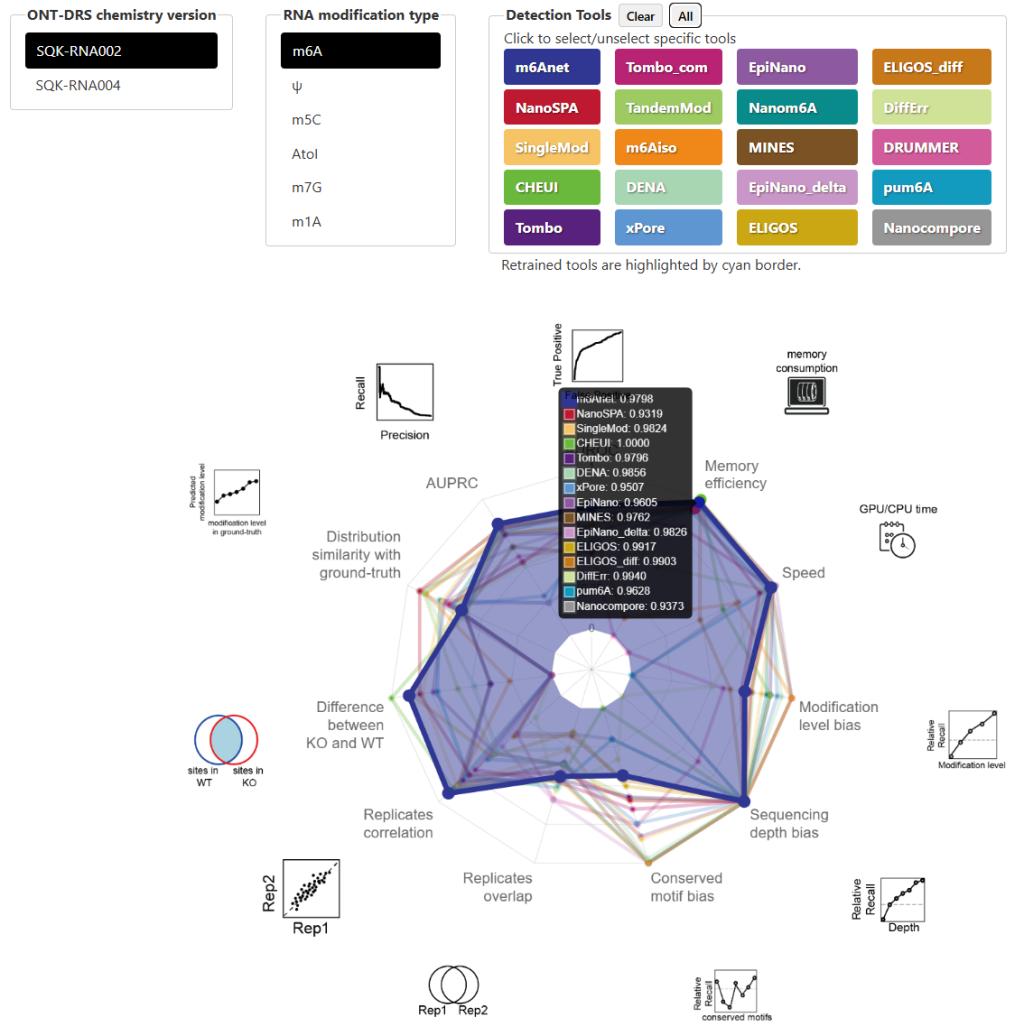
为促进领域发展,作者发布了全面的算法性能总结与在线资源平台NaRMBench,将每个工具的12项关键性能指标整合为交互式雷达图,方便用户根据需求进行选择与比较。该工作不仅为实验学者筛选适用分析工具提供了实用依据,也为算法开发指明了优化方向,为完善RNA修饰检测方法学奠定了重要的基准资源与权威指导。
同济大学生命科学与技术学院史偈君教授为论文通讯作者,课题组研究生罗婷婷与徐默萍为论文共同第一作者,研究生王淼、陈发迎亦对研究作出重要贡献。该研究获得国家自然科学基金资助。
论文链接:https://www.nature.com/articles/s41592-025-02974-y