生命科学与技术学院王晨飞团队揭示泛癌中广泛存在促纤维化生态位调控肿瘤免疫,成果发表于《自然肿瘤》
来源:生命科学与技术学院
时间:2025-08-27 浏览:
肿瘤是全球范围内导致人类死亡的主要原因之一。由于肿瘤微环境的不断进化及患者之间肿瘤微环境的高度异质性,只有少部分患者能够从免疫治疗和靶向治疗中受益。了解肿瘤进展过程中微环境的细胞组成变化,以及解析潜在促进其空间生态位产生的多细胞互作,对于准确靶向肿瘤微环境至关重要。目前,已有许多研究鉴定出在特定癌症中起抗肿瘤或促肿瘤作用的基质及免疫细胞类型。然而,由于癌症发生所在器官中组织驻留细胞的不同,不同癌症类型的肿瘤微环境中细胞状态和相互作用可能存在显著差异,阻碍了泛癌尺度有效的靶点开发。同时,领域内目前多数的泛癌分析往往关注于单一细胞类型,忽略了多细胞协作形成的空间结构对肿瘤发展及治疗效果的影响。

8月25日,同济大学生命科学与技术学院王晨飞团队在《自然肿瘤》(Nature Cancer)上发表了文章“Spatiotemporal analyses of the pan-cancer single-cell landscape reveal widespread profibrotic-associated ecotypes regulating tumor immunity”。该研究集成了来自公共数据集中36种癌症类型746个样本的超过400万个单细胞的数据,以及6种癌症类型62个样本的空间转录组学数据。涵盖癌旁正常组织、癌前病变组织、肿瘤组织和转移性肿瘤样本,并建立了系统描绘肿瘤微环境异质性的泛癌图谱TabulaTIME(http://wanglab-compbio.cn/TabulaTIME/)。研究人员对泛癌尺度肿瘤微环境内多个细胞谱系进行了详细注释,将其分为杀伤型淋巴细胞、调控型淋巴细胞、B淋巴细胞、髓系细胞、成纤维细胞及内皮细胞六个大的细胞谱系,探讨了这些细胞亚型与预后的关系,识别出在肿瘤发生和进展过程中起重要作用的细胞亚型,并对肿瘤特异的细胞类型和多细胞互作进行了深度分析(图1)。
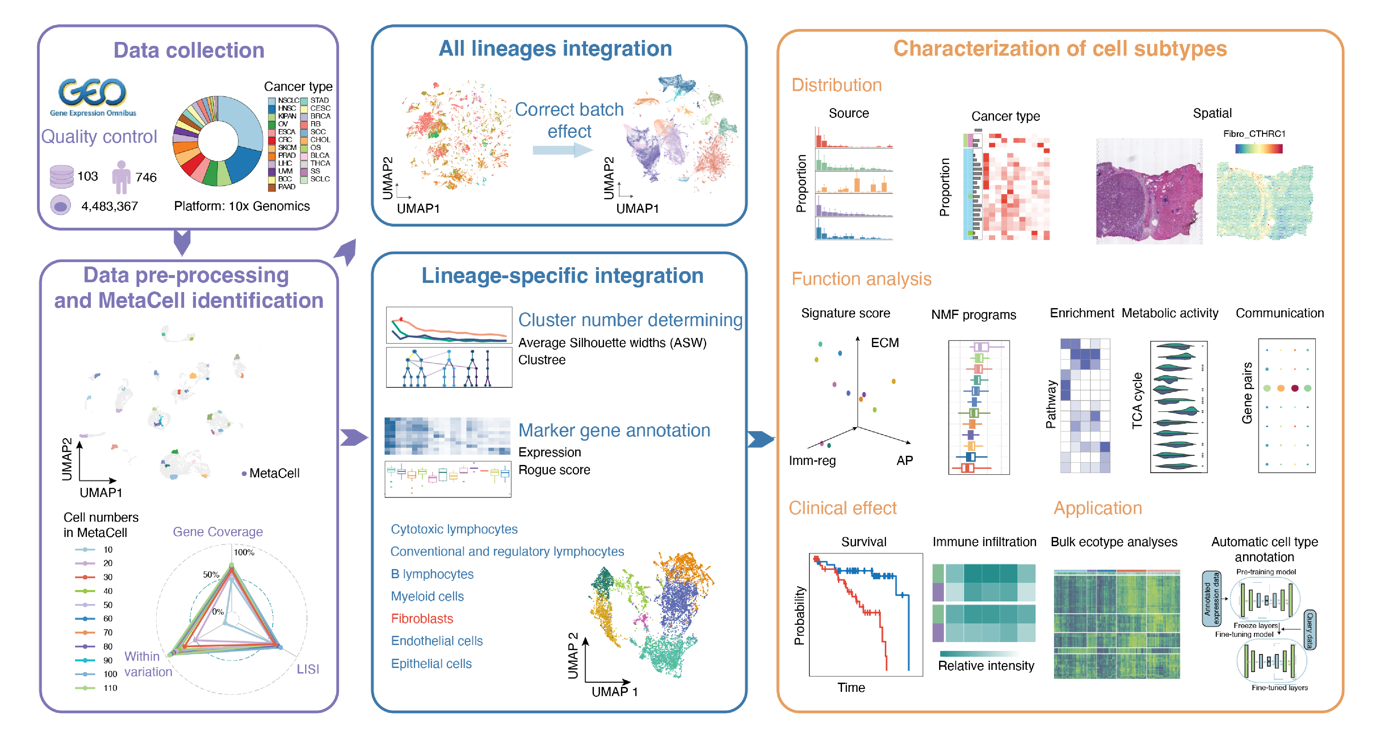
图1 TabulaTIME研究框架
基于TabulaTIME框架,研究人员发现CTHRC1阳性的成纤维细胞高表达细胞外基质重塑相关基因,并广泛存在于多种原发肿瘤样本中,但在正常组织中极少出现。结合空间转录组学数据及三种癌症类型的免疫荧光染色,研究人员发现CTHRC1阳性的成纤维细胞富集于恶性细胞与正常组织之间的边界区域,形成了一层包裹在肿瘤细胞边界上的免疫屏障。SLPI阳性的巨噬细胞同样高表达细胞外基质重塑相关基因,表现出促纤维化的表型。在泛癌尺度,CTHRC1阳性成纤维细胞与SLPI阳性巨噬细胞在肿瘤组织中呈现明显的共定位,形成了促纤维化的生态位调节肿瘤免疫,且与肿瘤的不良预后显著相关。最后,该研究还证明了TabulaTIME图谱可以用于解析大规模队列数据中患者微环境中的细胞组成,并将不同癌症类型的样本根据微环境特征分为五种不同的肿瘤生态系统。研究还证明,TabulaTIME注释过的图谱可以作为参考集,用于训练基于深度学习的细胞自动注释方法,准确有效地注释肿瘤相关的单细胞转录组数据集(图2)。综上,该项研究通过整合泛癌尺度的单细胞及空间组学公共数据,阐明了泛癌尺度存在的肿瘤特异细胞类型及互作关系,为准确靶向肿瘤空间生态位进行疾病干预提供了重要的研究视角及潜在靶点,也为大规模多模态数据整合建立了新的计算生物学框架。
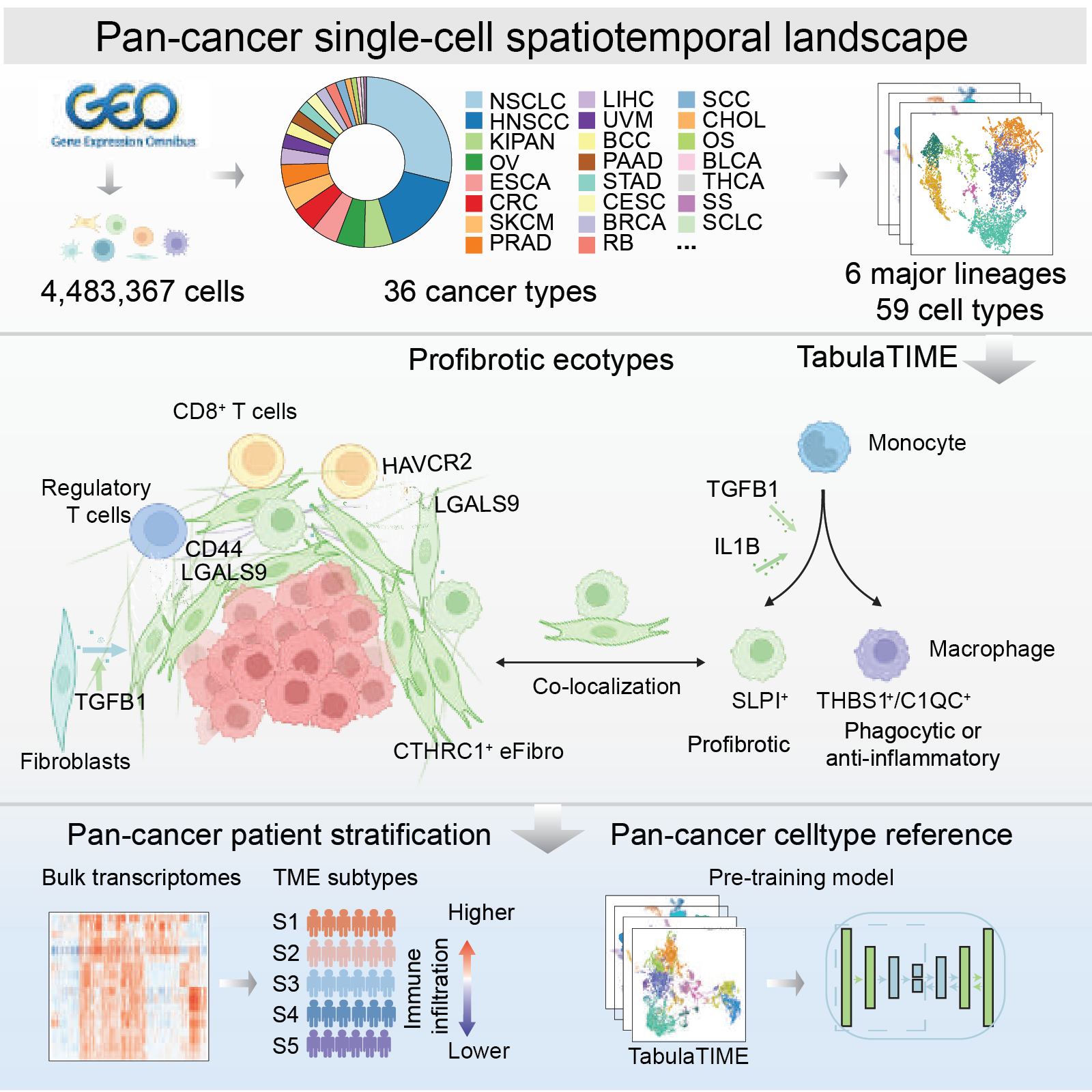
图2 TabulaTIME框架的主要发现和下游应用
同济大学生命科学与技术学院/附属同济医院/上海自主智能无人系统科学中心王晨飞教授和同济大学生命科学与技术学院吴秋助理教授为论文共同通讯作者,博士后韩雅为论文第一作者。该研究得到了同济大学附属上海市肺科医院张鹏教授、张乐乐副研究员,附属东方医院李芳教授以及四川大学华西口腔医院李太文副研究员的大力支持。
论文链接:https://www.nature.com/articles/s43018-025-01039-5