生命科学与技术学院张家民、高绍荣、史偈君、刘晓雨团队合作开发cFOOT-seq:单分子、单细胞分辨率的转录因子占位检测新平台,成果发表于《蛋白与细胞》
来源:生命科学与技术学院
时间:2025-08-11 浏览:
在基因调控的复杂网络中,转录因子(Transcription Factor, TF)就像细胞的“指挥家”,通过协同调控启动子、增强子和染色质状态,驱动细胞命运、发育过程和应激响应。真正理解这场分子“协奏曲”,不仅要知道单个TF的作用,还需要同步描绘数百种TF的结合模式及其所处的染色质环境。然而,现有技术要在一次实验中同时获取这些信息几乎不可能,这成为表观遗传与转录调控研究的核心瓶颈。
近日,同济大学生命科学与技术学院张家民教授、高绍荣教授、史偈君教授、刘晓雨教授多个课题组团队合作,在《蛋白与细胞》(Protein&Cell)发表研究论文“Genome-Wide Investigation of Transcription Factor Footprints and Dynamics Using cFOOT-seq”,报道了全新的cFOOT-seq技术。这一方法通过高效、低序列偏好的双链DNA脱氨酶SsdAtox,在全基因组范围内以单碱基分辨率同时检测染色质可及性、核小体定位和数百种TF足迹,并可与ATAC-seq联用,实现单分子乃至单细胞层面的定量分析,突破了传统方法在多靶点检测和灵敏度上的限制。

核心亮点一:高效SsdAtox酶,精准原位标记
研究团队比较了多种双链DNA脱氨酶的效率和特性,最终选择SsdAtox作为核心探针。它能高效将开放染色质上的胞嘧啶(C)脱氨为尿嘧啶(U),测序后转化为胸腺嘧啶(T),从而直接“写入”染色质可及性、核小体分布和TF结合位点信息。一次实验即可解析上百种TF的占位信息,适用于开放区与非开放区,且对低起始量样本同样适用。
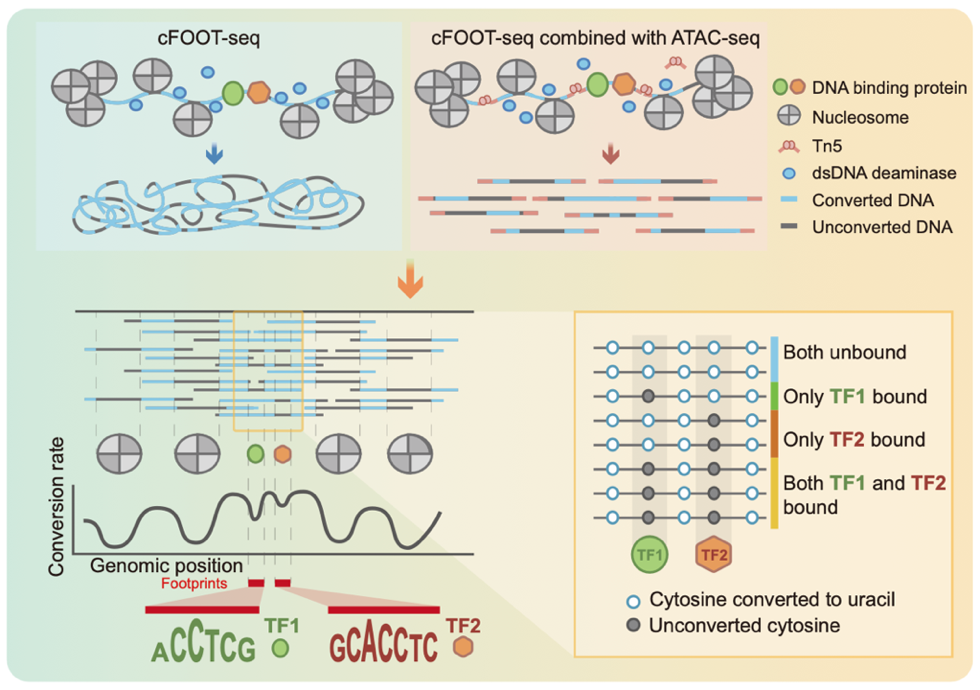
核心亮点二:多维度协同检测,构建高分辨率染色质景观
cFOOT-seq将TF结合信息与染色质状态信息融合在同一测序框架中,提供多维协同的“染色质景观”图谱。这使研究者能够在单次实验中,不仅看到“谁”在结合,还能理解TF与其染色质环境如何互动,为解析多TF协同调控和动态行为提供了坚实数据基础。
核心亮点三:联用ATAC-seq,进军单分子与单细胞
cFOOT-seq保留DNA完整性,天然适合与其他组学方法联用。与ATAC-seq结合,形成ATAC-cFOOT-seq(开放区富集更强)和cFOOT-ATAC-seq(检测灵敏度更高)两种策略,不仅降低测序成本,还可定量分析单分子TF占位及协同关系。结合孔板建库流程,团队开发出scATAC-cFOOT-seq,将TF占位检测拓展到单细胞分辨率,为研究复杂组织和异质性细胞群体提供了强有力的工具。
核心亮点四:FootTrack智能分析平台
配套的FootTrack平台利用FOS(Footprint Occupancy Score)和TFOS(Transcription Factor Occupancy Score)对TF结合强度进行精确量化,并支持基于motif的 de novo足迹预测,帮助发现新的TF调控因子和模式。
科研应用:捕捉转录因子的动态足迹
在OSKM诱导重编程、SWI/SNF染色质重塑抑制/恢复等模型中,cFOOT-seq成功捕捉到关键TF的动态变化。在BRM014处理的HepG2细胞中,该方法揭示了具有相似SWI/SNF依赖性的TF往往在基因组上空间邻近,暗示着一种高效的染色质重塑与TF结合布局模式。
cFOOT-seq融合高分辨率、定量能力与高通量优势,将TF结合与染色质状态“一体化”编码入DNA序列中,为精准解析基因调控机制、揭示调控“语法”以及构建动态基因调控网络提供了全新的技术平台。未来,它有望广泛应用于发育生物学、再生医学、疾病机制研究等领域,并推动表观遗传学与转录调控研究迈入多维整合的新阶段。
同济大学生命科学与技术学院博士研究生王姮、武昂和杨孟臣为论文共同第一作者。同济大学生命科学与技术学院张家民教授、高绍荣教授、史偈君教授与刘晓雨教授为论文共同通讯作者。该研究由同济大学附属同济医院教育部脊髓损伤与修复重点实验室、同济大学生命科学与技术学院、同济大学附属第一妇婴保健院、教育部“细胞干性与命运编辑“前沿科学中心共同完成,并获得国家重点研发计划、国家自然科学基金和同济大学与附属同济医院启动经费等项目的支持。
论文链接:https://academic.oup.com/proteincell/advance-article/doi/10.1093/procel/pwaf071/8224573?searchresult=1